User:SFLSNZYN/工作区/20

CRISPR(IPA:/ˈkrɪspər/;DJ:/ˈkrispə/;KK:/ˈkrɪspɚ/)是存在於细菌中的一種基因組,該類基因組中含有曾經攻擊過該細菌的病毒的基因片段。細菌透過這些基因片段來偵測並抵抗相同病毒的攻擊,並摧毁其DNA。這類基因組是細菌免疫系统的關鍵組成部分。透過這些基因组,人類可以準確且有效地編輯生命體内的部分基因,也就是CRISPR/Cas9基因編輯技術。
CRISPR/Cas系統,為目前發現存在於多數細菌與絕大多數的古菌中的一種後天免疫系統[2],以消滅外來的質體或者噬菌體[3][4],並在自身基因組中留下外來基因片段作為“記憶”[5]。全名為常間回文重複序列叢集/常間回文重複序列叢集關聯蛋白系統(Clustered Regularly Interspaced Short Palindromic Repeats/CRISPR-associated proteins)。
目前已發現三種不同類型的 CRISPR/Cas系統,存在於大約40%和90%已定序的细菌和古菌中[6][7]。其中第二型的組成較為簡單,以Cas9蛋白以及嚮導RNA(gRNA)為核心的組成。
Cas9是第一個被廣泛應用的CRISPR核酸酶,其次是Cpf1,其在新澤西弗朗西斯菌的CRISPR/Cpf1系统中被發現[8][9]。其它這樣的系統被認為存在[10]。
由於其對DNA干擾(DNAi)的特性(參見RNAi),目前被積極地應用於遺傳工程中,作為基因體剪輯工具,與鋅指核酸酶(ZFN)及類轉錄活化因子核酸酶(TALEN)同樣利用非同源性末端接合(NHEJ)的機制,於基因體中產生脱氧核醣核酸的雙股斷裂以利剪輯。二型CRISPR/Cas並經由遺傳工程的改造應用於哺乳類細胞及斑馬魚的基因體剪輯[11][12]。其設計簡單以及操作容易的特性為最大的優點。未來將可應用在各種不同的模式生物當中。
| Cascade (CRISPR相關病毒防禦複合體) | |
|---|---|
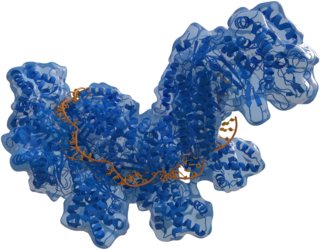
| |
| Structure of crRNA-guided E. coli Cascade complex (Cas, 藍色) bound to single-stranded DNA (橘色). | |
| 标识 | |
| 生物 | |
| 符号 | ? |
| PDB | 4QYZ |
| 其他数据 | |
發現歴史
编辑聚簇DNA重複的發現始於世界三個地區的三個獨立地點。我們今天稱為CRISPR的基因組重複群集,即原核生物擬核DNA鏈中的丛生重复序列,在1987年一份由大阪大学的石野良純領銜的大腸桿菌研究报告中被首次描述[13]。2000年,相似的重复序列在其它真细菌和古细菌中被发现,并被命名为短间隔重复序列(Short Regularly Spaced Repeats,SRSR)[14]。2002年SRSR被重命名为CRISPR,其中一部分基因编码的蛋白为核酸酶和解旋酶,这些关联蛋白(Cas, CRISPR-associated proteins)与CRISPR组成了CRISPR/Cas系统[15]。
Cas9
编辑科学家还研究了来自化脓性链球菌的更简单的CRISPR系统,其依赖于蛋白Cas9。Cas9内切核酸酶是包含两个小RNA分子的四组分系统[16]。
詹妮弗·杜德纳、埃马纽埃尔·卡彭蒂耶及張鋒各自独立的探索CRISPR关联蛋白,了解细菌如何在它们的免疫防御使用间隔(spacer)。她们共同研究一个比较简单的依赖于称为Cas9蛋白质的CRISPR系统。
Cpf1
编辑在2015年,核酸酶Cpf1被发现在新泽西弗朗西斯菌的CRISPR/Cpf1系统[8][9]。其他这样的系统被认为存在[10]。Cpf1显示了与Cas9的几个关键差异,包括:导致双链DNA中的“交错”切割,而不是由Cas9产生的“钝的”切割,依赖于“富含T碱基”的原间隔序列(Protospacer)相邻基序(为Cas9提供替代的靶向位点) 仅需要CRISPR RNA(crRNA)用于成功靶向(使用Cas9同时需要crRNA和一个反式激活的crRNA(tracrRNA))。
評價
编辑在2012年和2013年,CRISPR是《科学杂志》年度突破的第二名。在2015年,它是该奖项的赢家。CRISPR在2014年和2016年被《麻省理工科技評論》评为10项突破技术之一[17][18]。
参阅
编辑參考文獻
编辑- ^ Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea. Science. January 2010, 327 (5962): 167–70. PMID 20056882. doi:10.1126/science.1179555.
- ^ Westra ER, Swarts DC, Staals RH, Jore MM, Brouns SJ, van der Oost J. The CRISPRs, they are a-changin': how prokaryotes generate adaptive immunity. Annu Rev Genet. 2012, 46: 311–339. PMID 23145983.
- ^ Barrangou R, Fremaux C, Deveau H; et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science. March 2007, 315 (5819): 1709–12. PMID 17379808. doi:10.1126/science.1138140.
- ^ Marraffini LA, Sontheimer EJ. CRISPR Interference Limits Horizontal Gene Transfer in Staphylococci by Targeting DNA. Science. December 2008, 322 (5909): 1843–5. PMC 2695655 . PMID 19095942. doi:10.1126/science.1165771.
- ^ Marraffini LA, Sontheimer EJ. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nat Rev Genet. February 2010, 11 (3): 181–190. PMC 2928866 . PMID 20125085. doi:10.1038/nrg2749.
- ^ 71/79 Archaea, 463/1008 Bacteria CRISPRdb, Date: 19.6.2010
- ^ Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats. BMC Bioinformatics. 2007, 8: 172. PMC 1892036 . PMID 17521438. doi:10.1186/1471-2105-8-172.
- ^ 8.0 8.1 Zetsche B, Gootenberg JS, Abudayyeh OO, Slaymaker IM, Makarova KS, Essletzbichler P, Volz SE, Joung J, van der Oost J, Regev A, Koonin EV, Zhang F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. October 2015, 163 (3): 759–71. PMID 26422227. doi:10.1016/j.cell.2015.09.038.
- ^ 9.0 9.1 Fonfara I, Richter H, Bratovič M, Le Rhun A, Charpentier E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. April 2016, 532 (7600): 517–21. PMID 27096362. doi:10.1038/nature17945.
- ^ 10.0 10.1 Even CRISPR. The Economist. [2016-05-25]. ISSN 0013-0613.
- ^ Mali P, Yang L, Esvelt KM, Aach J, Guell M, Dicarlo JE, Norville JE, Church GM. RNA-Guided Human Genome Engineering via Cas9. Science. 2013. PMID 23287722.
- ^ Hwang WY, Fu Y, Reyon D, Maeder ML, Tsai SQ, Sander JD, Peterson RT, Yeh JR, Joung JK. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nat Biotechnol. 2013. PMID 23360964. doi:10.1038/nbt.2501.
- ^ Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J Bacteriol. 1987, 169 (12): 5429–33. PMC 213968 . PMID 3316184.
- ^ Mojica FJM, Díez-Villaseñor C, Soria E, Juez G. Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria. Mol Microbiol. 2000, 36 (1): 244–6. PMID 10760181. doi:10.1046/j.1365-2958.2000.01838.x.
- ^ Jansen R, Embden JD, Gaastra W, Schouls LM. Identification of genes that are associated with DNA repeats in prokaryotes. Mol Microbiol. 2002, 43 (6): 1565–75. PMID 11952905. doi:10.1046/j.1365-2958.2002.02839.x.
- ^ Barrangou R. Diversity of CRISPR-Cas immune systems and molecular machines. Genome Biology. November 2015, 16: 247. PMC 4638107 . PMID 26549499. doi:10.1186/s13059-015-0816-9.
- ^ Talbot D. Precise Gene Editing in Plants/ 10 Breakthrough Technologies 2016. Massachusetts Institute of Technology. 2016 [18 March 2016].
- ^ Larson C, Schaffer A. Genome Editing/ 10 Breakthrough Technologies 2014. Massachusetts Institute of Technology. 2014 [18 March 2016].